Rädler Group
We are a soft matter physics group studying self-assembly and dynamics of macromolecules and lipid membranes and the spatio-temporal self-organization of living cells (active matter).
We are a soft matter physics group studying self-assembly and dynamics of macromolecules and lipid membranes and the spatio-temporal self-organization of living cells (active matter).

Join the Rädler Group's weekly seminar featuring invited speakers, primarily from the life sciences, sharing insights into cutting-edge research. 🗓️ Wednesdays, 4 p.m. during the lecture period.
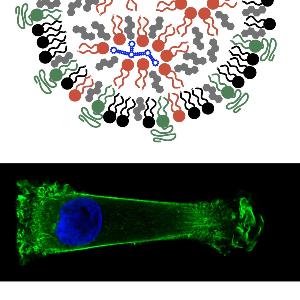
In gene and siRNA based therapy, therapeutic benefits rely on the efficient transfer of foreign nucleic acids into cells. We investigate synthetic complexes capable of readily carrying nucleic acid across the cell membrane. The self-assembly, size and structure of lipid- and polycation-DNA complexes, known respectively as lipoplexes and polyplexes, are studied. We also endeavor to quantify and model transfection at the single cell level using GFP reporter and single cell time-lapse microscopy.
Read more
Collective cell migration is a key element in morphogenesis, cancer formation and wound healing. The multicellular dynamics of epithelial cell layers were studied in experiment and theory over the last years. Yet little is known about cell patches of finite size that should lead over from single cell behavior to collective motion. Here we study the migration and proliferation of small assemblies of epithelial cells.
Read more
We are interested in the kinetics of cellular responses, in particular in gene expression after transfection and event cascades in cytotoxicity. Using live cell imaging on single cell arrays (LISCA) time courses from individual cells are monitored and analyzed based on mathematical models. Cellular microarrays help to resolve cellular heterogeneity, a hallmark of complex biological systems dominated by intrinsic fluctuations.
Read more

We fabricate artificially designed micro-environments to culture and probe living cells. Arrays of defined cavities provide standardized geometric boundaries for individual cells or groups of cells. We study single cell gene expression, cell migration and apoptotic events with precise spatio-temporal control. To this end we develop biofabrication techniques, including photo-lithography, hot-embossing, electroplating, soft lithography, 3D printing and microcontact printing, to create two-dimensional (2D) and three-dimensional (3D) pattern, cavities and channels made of biocompatible materials like PDMS, COC, hydrogels and proteins. The 3D microenvironments allow for long-term observation of growth and function of individual living cells, interacting groups of cells and organoids.
Supported membranes are versatile biomimetic interfaces. We study the structure and dynamics of supported membranes on various solid supports in collaboration with the group of Bert Nickel. The control of fluidity, spacing and lateral organisation is a current research challenge.
Find a list of all publications by the Rädler Group on this page.
We are offering a variety of projects for bachelor, master and PhD theses.
| Name | Position |
|---|---|
| Rädler, Joachim | |
| Meixner, Margarete | Administration |
| Leu, Charlott | Technical Staff |
| Schwake, Gerlinde | Technical Staff |
| Rüter-Stimpfle, Margarita | Administration |
| Jouneau, Agathe | PhD Student |
| Philipp, Julian | PhD Student |
| Maksutova, Daria | PhD Student |
| Stockbauer, Simon | Master student |
| Kostyurina, Ekaterina | Postdoc |
| Kirchmair, Bernhard | PhD Student |
| Kuhl, Santiago | PhD Student |